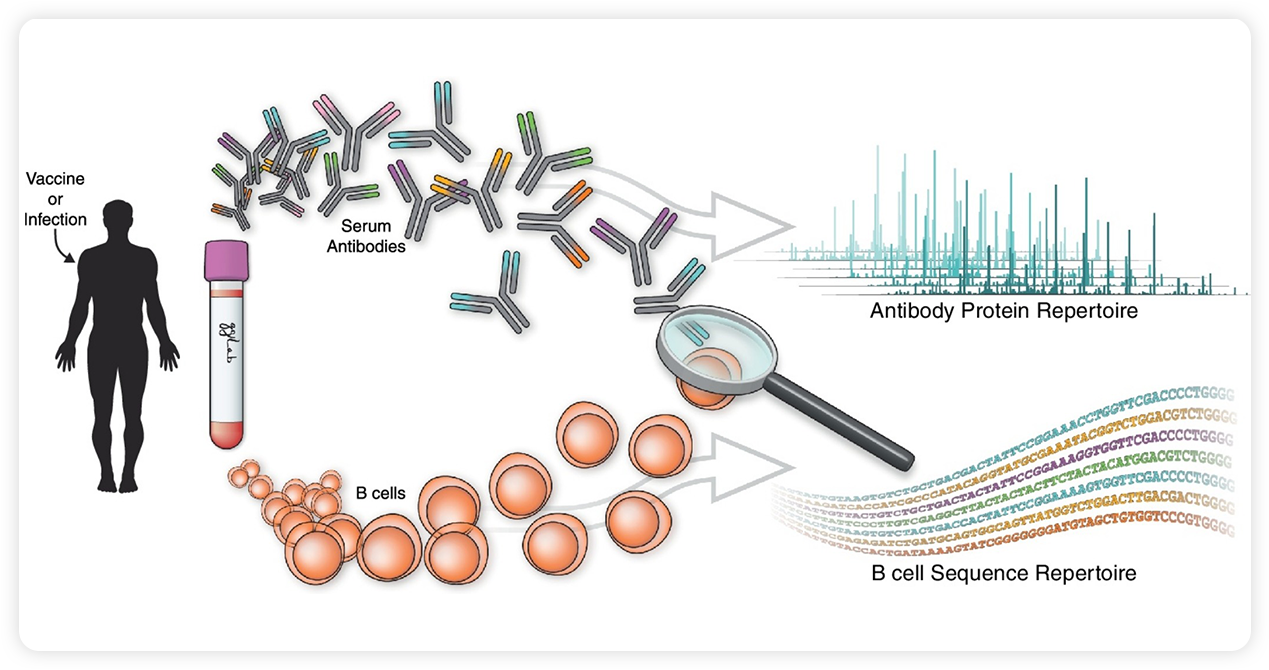
噬菌体免疫沉淀测序(Phage immunoprecipitation sequencing, PhIP-seq)技术是近年来新兴的一项高通量筛选的技术,联合高容量的噬菌体展示多肽库、抗体免疫沉淀以及高通量测序技术,可用于分析体液(如血清)中抗体所识别的抗原组成,基于抗原-抗体匹配的、全局性、系统性的抗体库(Antibody repertoire)分析,是抗体组学(Antibodyomics)研究的有力工具。
抗码生物的创始团队于2015年搭建了PhIP-seq平台(Wu FL, MCP, 2019; Qi H, MCP, 2021),经过多年的技术沉淀、改进和优化,形成了成熟、稳定、可靠的PhIP-seq的实验平台,制备了包含人类蛋白质组、细菌/病毒蛋白质组等多种噬菌体展示肽库,并联合多肽芯片验证,形成了一整套抗体组学解决方案。
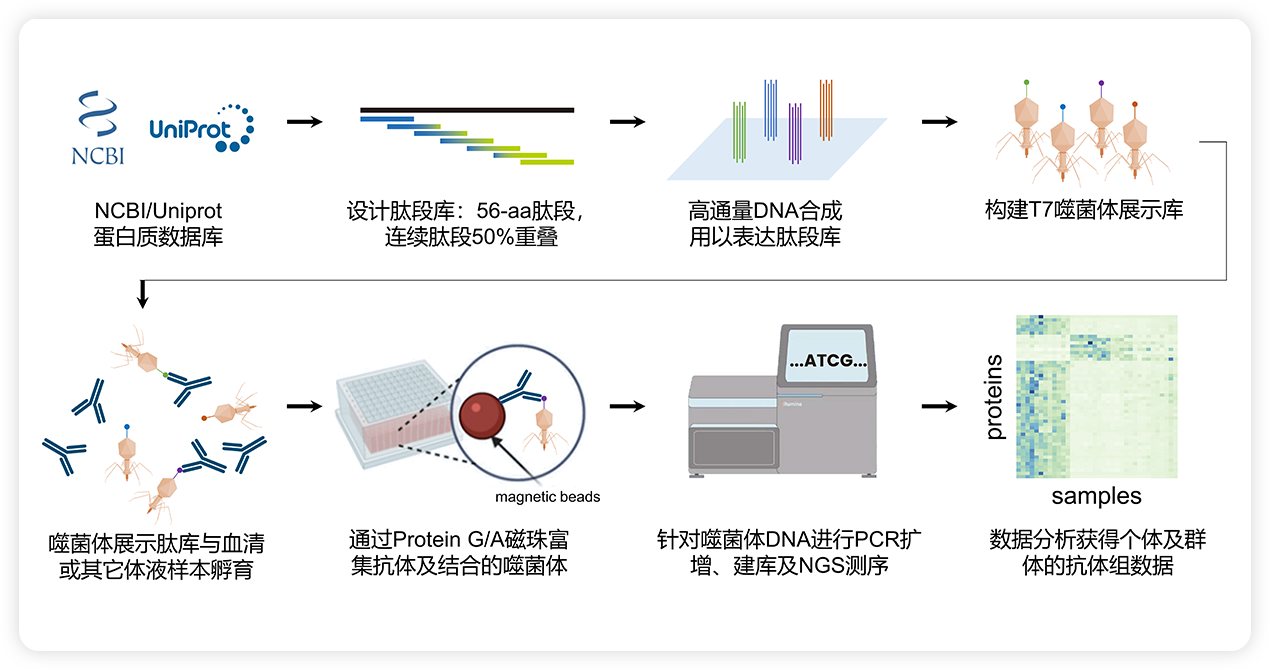
PhIP-seq技术可分为两部分:
根据应用需求建立抗原库。抗原库为噬菌体展示的多肽库。多肽序列来自公开的蛋白质数据库,如NCBI或Uniprot。根据需要可针对人、小鼠、各类细菌、各类病毒以及过敏原的蛋白质或蛋白质组进行设计,通常可覆盖上万种蛋白质,对应数十万条肽段。针对所设计的多肽序列,反向翻译为DNA序列,进行密码子优化,并采用高通量合成的方法获得DNA文库。最终,将DNA文库构建载体并包装到T7噬菌体,获得噬菌体展示多肽库。所获得噬菌体展示库须经过深度测序以评估其质量。
样本筛选和数据分析。将含有抗体的样本(如血清、血浆、脑脊液等)与噬菌体展示多肽库共孵育,并通过免疫沉淀的方式富集抗体-抗原(噬菌体)复合物。针对富集的噬菌体进行建库、高通量测序、序列解析,即可获得每一样本中抗体所识别的多肽序列的集合。通过整合分析可进一步获得蛋白抗原的信息。
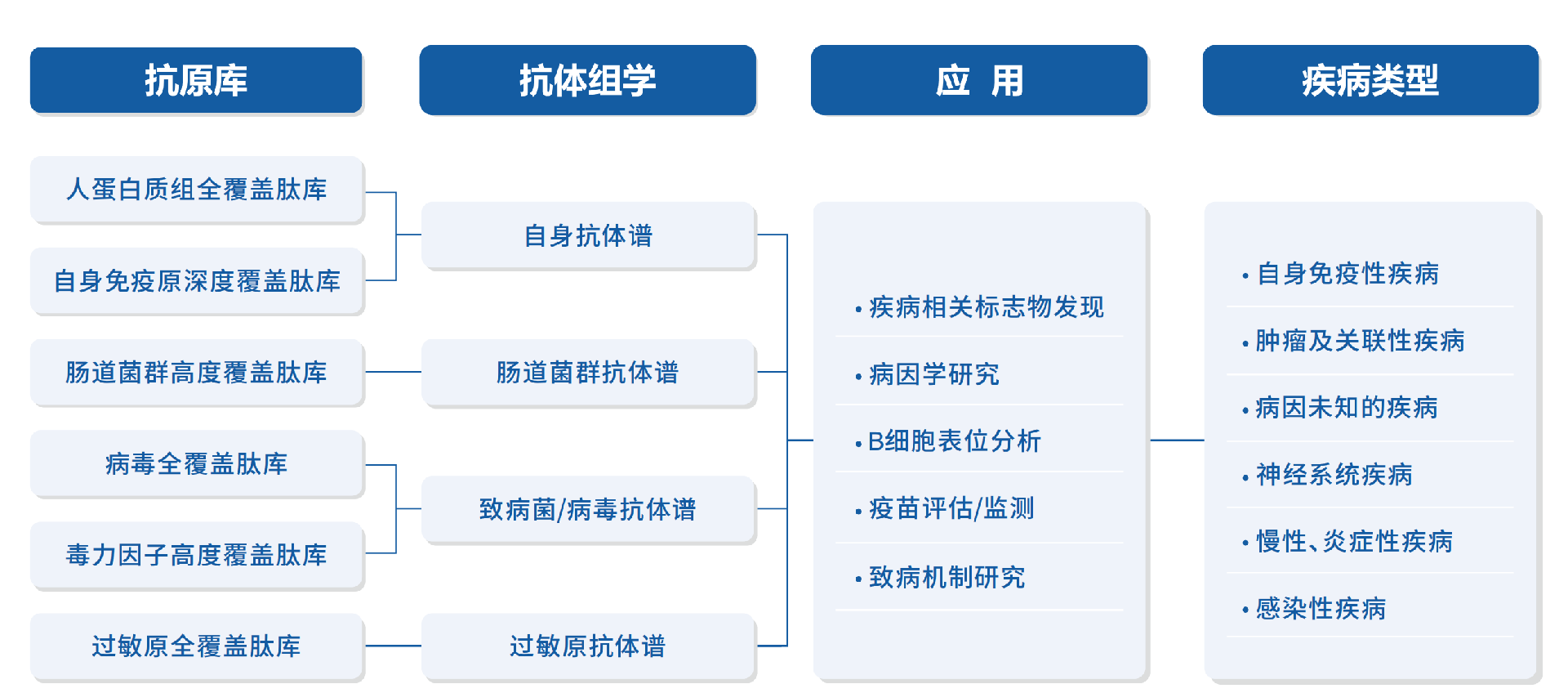
已经建立的抗原库
人蛋白质组全覆盖 病毒全覆盖 毒力因子高度覆盖
涵盖人类参考基因组>5万个蛋白, 涵盖260种人类病毒的9,831个蛋白, 涵盖各类致病菌的~14,186个毒力因子蛋白,
包含~60万条肽段。 包含>12万条肽段。 包含>17万条肽段。
肠道菌群高度覆盖 自身免疫原深度覆盖 过敏原覆盖
涵盖>800个肠道菌类型,对应 针对92种自免病的>720个自免 包含856个物种的5,436个过敏原蛋白质,
对应>20,000个分泌蛋白、膜蛋白, 蛋白抗原深度覆盖,包含~36万条肽段。 共计>3.8万条肽段。
包含>24万条肽段。
应用场景
血清/血浆;
脑脊液;
肺泡灌洗液
……

