文章解析∣Immunity背靠背:噬菌体免疫沉淀测序技术(PhIP-seq)解析抗体组
噬菌体免疫沉淀测序(Phage immunoprecipitation sequencing, PhIP-seq)技术是近年来新兴的一项高通量筛选的技术,联合高容量的噬菌体展示多肽库、抗体免疫沉淀以及高通量测序技术,可进行基于抗原-抗体匹配的、全局性、系统性的抗体库(Antibody repertoire)分析。该技术灵敏度高、可以高通量检测抗体与多肽的结合,通过研究结合位点深入解析抗体与靶标的作用机制,广泛用于研究自身抗体组研究、感染性疾病相关抗体以及疫苗研发等领域,为解析复杂的免疫学问题提供了有力工具,在生物医学研究中有重要应用价值。
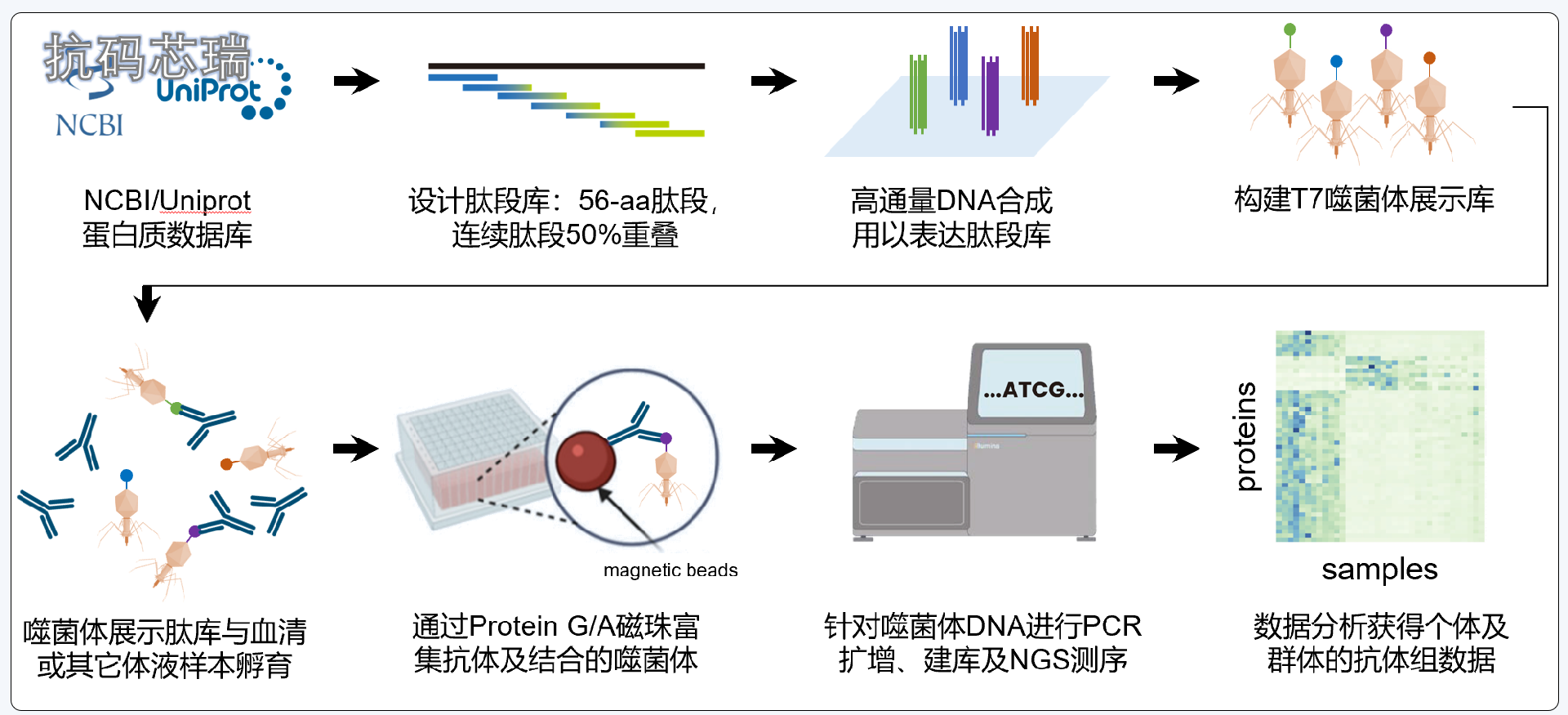
PhIP-seq技术原理流程
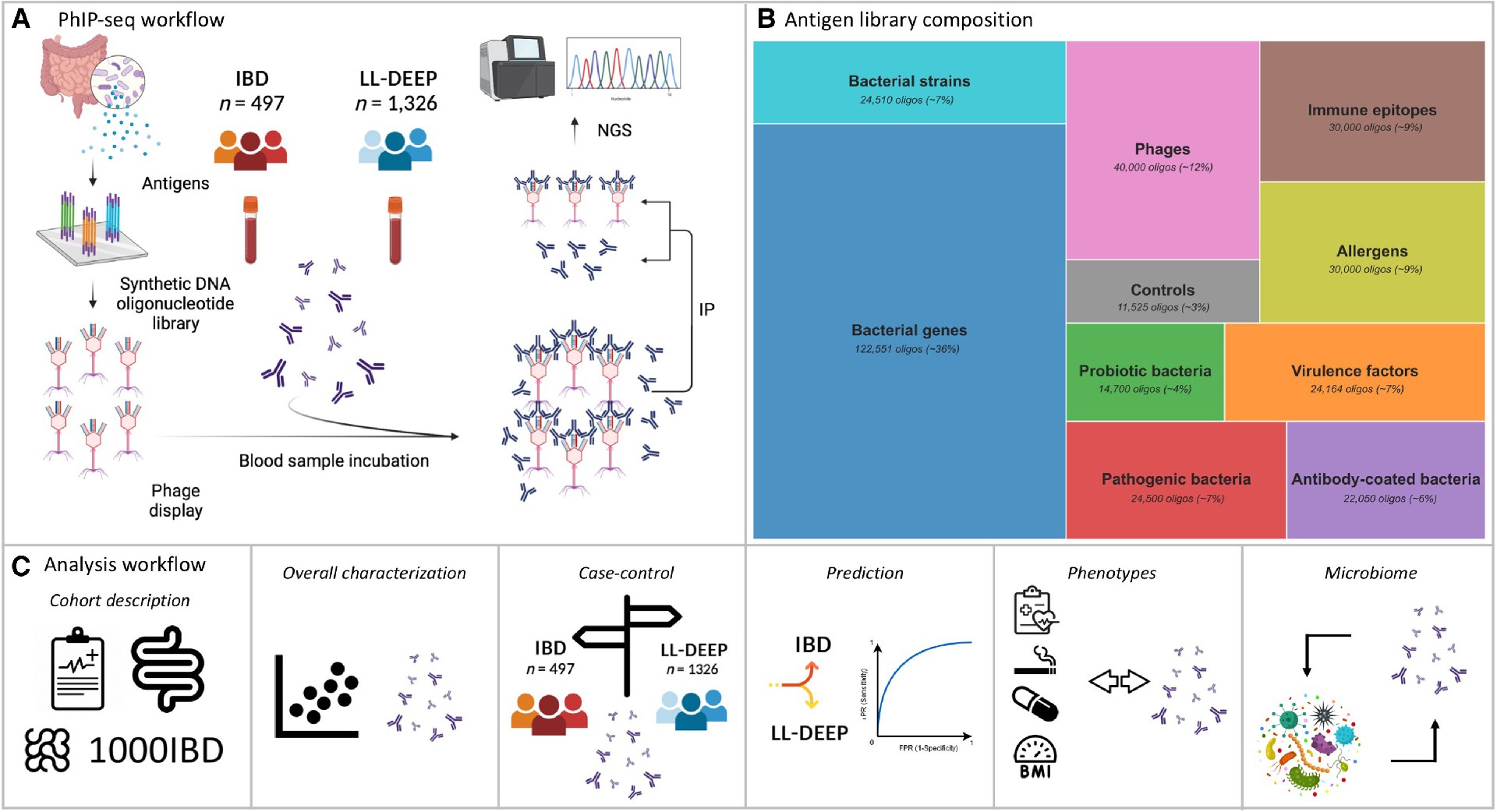
近期,来自荷兰格罗宁根大学的研究团队在免疫学顶刊Immunity上背靠背发表了两篇文章,基于噬菌体免疫沉淀检测(PhIP-seq)技术深入构建和分析了人血清的抗体库组成,并发现了炎症性肠病(IBD)的特征抗体标志物。这两篇文章采用了相同的噬菌体展示肽库,筛选了超过1000例血清样本,从不同角度进行了深入分析。该噬菌体展示肽库包含344,000条多肽,其对应的蛋白来源于微生物(肠道微生物、益生菌和致病菌等)、致病因子数据库(VFDB)、免疫表位数据库(IEDB)。每条肽的单个长度为64个氨基酸(两条相邻肽重叠部分为20个氨基酸)。

第一篇文章聚焦于炎症性肠病(IBD)(包括Crohn病(CD)和溃疡性结肠炎(UC))病人的抗体组分析。研究者采用高通量噬菌体展示免疫沉淀测序(PhIP-seq)技术,检测了497例IBD患者与1326例对照者的血清抗体,通过对比分析发现炎症性肠病患者的抗体表位谱与健康对照有明显差异,获得了373种IBD差异表达的抗体反应(202种过度表达和171种低表达),其中17%同时存在于IBD的两种形式,55%仅存在于CD,28%仅存在于UC。这些多肽之间存在一定的共线性,提示它们可能具有共同的抗体识别基序或结构域。
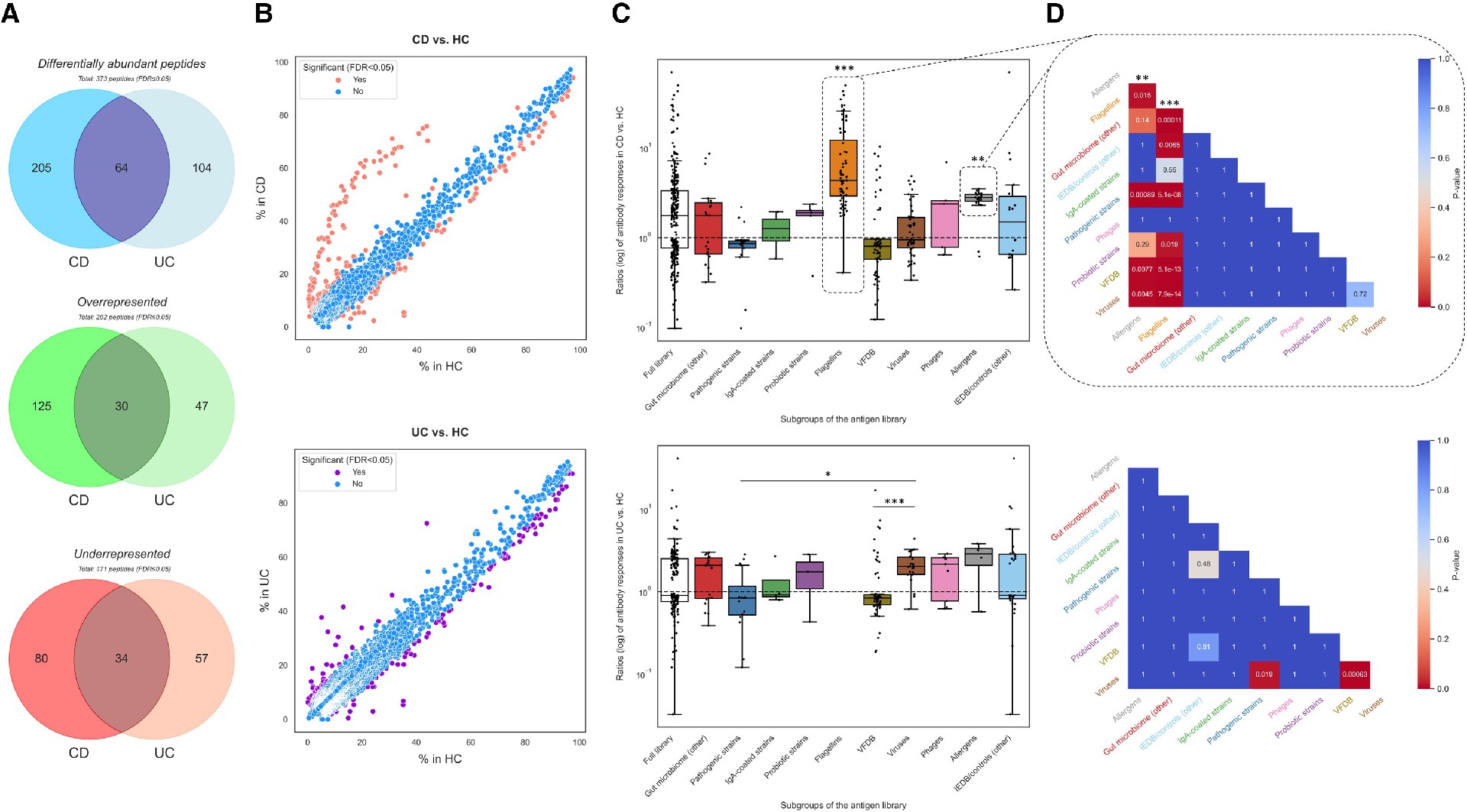
IBD患者与正常人的抗体表位图谱存在明显差异
其次,研究还评估了这些多肽与患者临床特征的相关性,,发现与疾病活动度、炎症指标和并发症存在相关,其对应抗体可能源于发病前便已存在的遗传易感性,在发病后被激活或被重新定向。
接着,作者选取少量差异化的肽段,采用递归特征消除(RFE)来建立和优化模型,用以区分IBD和对照样本。通过拟合,抗体表位谱可以准确区分克罗恩病患者(ID)和对照(曲线下面积[AUC] = 0.89),仅使用10个抗体也可较为准确的区分(AUC = 0.87)。表明这些特异性的多肽可以用于IBD的诊断。
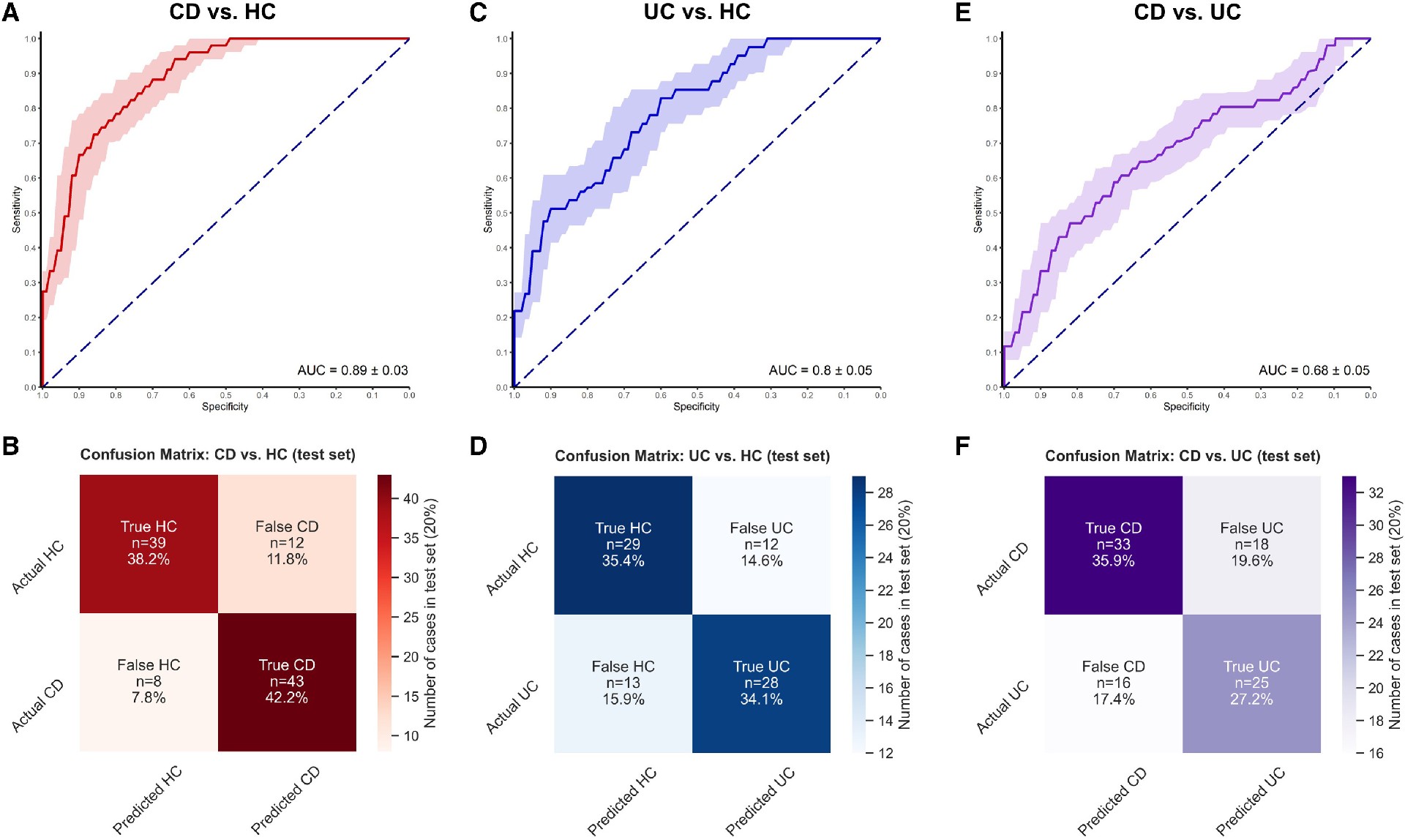
根据抗体表位谱对不同组别进行分类
进一步分析,发现针对细菌鞭毛素的抗体反应在CD中占主导地位,,且与回肠病变、纤维增生狭窄性疾病和抗酵母菌抗体阳性相关,但与粪便微生物群落构成无关。IBD患者抗体反应表现出选择性,特别是CD患者针对微生物鞭毛的反应。鞭毛是CD的主要抗原,可以引起强烈的IgG免疫应答。研究者发现许多针对Lachnospiraceae鞭毛(例如Roseburia、Clostridium、Eubacterium和Agathobacter属的鞭毛)的抗体应答,特别是在(回肠)CD患者中。这些结果证实了以前针对IBD自适应免疫应答的研究结果。这种抗鞭毛抗体特征对CD而不是UC的特异性强调了这两种疾病的病理生理学不同,这可能由CD特有的免疫(例如异常Th1驱动免疫)或遗传易感性解释,可以追随这一原理设计和测试针对鞭毛的免疫治疗。
综上,该研究采用PhIP-seq技术分析了IBD病人的抗体表位谱,发现了多种与疾病相关的特异抗体,可作为诊断指标,并提供了肠道微生物的异常免疫反应和IBD致病机制的进一步证据。以上结果说明PhIP-Seq数据与其他生物信息层面的结合可以改善我们对IBD的分子理解,并提供机制研究的线索。同时,PhIP-Seq或可成为一种有价值的临床应用,捕捉宿主适应性免疫与肠道微生物之间的界面,为IBD患者创建“免疫指纹”。

另一篇文章则基于PhIP-seq对环境因素和遗传学因素与人体适应性免疫影响的关系进行了深入剖析。
首先,基于相似的数据集(主要是健康对照组),作者发现抗体谱具有显著的个体差异,家庭成员之间的相似度相对更高。遗传学分析显示HLA、IGHV和FUT2基因区域与抗体结合多肽的反应活性有关,例如通过推断HLA等位基因、氨基酸和结构变异,确定了500多个多肽存在与特定HLA的关联。同时,通过探索多肽库与各种形态学、生活方式因素之间的关系,观察到EBV和CMV与淋巴细胞和中性粒细胞计数相关,这些发现与EBV或CMV感染患者检查结果中的绝对淋巴细胞增多和中性粒细胞减少相一致。来自EBV和识别为与EBV共存的一组多肽的抗体结合多肽在女性中也更为普遍,这可能导致更高的致病率。接着,研究评估了影响抗体表位谱的因素,发现遗传因素可以解释61%的变异,环境暴露史可以解释6%的变异。还有一部分变异可能源于人体内在的随机过程。研究还发现,抗体表位谱与人群特征如性别、年龄、居住环境也存在一定相关,其中居住在农村的研究对象识别的微生物多肽更丰富。最后,研究评估了每个人5年内抗体表位谱的稳定性,发现大部分抗体为稳定存在的抗体,,但新出现的抗体也可占到总抗体的30%。总之,该研究聚焦于人群的抗体表位谱的特征与变异,,发现抗体谱存在较高的个体差异也存在有一定的遗传相关性。研究评估了多重因素对这些差异的作用,包括遗传因素、环境因素和人体内在随机过程。
综上, PhIP-seq技术可进行基于抗体-抗原匹配分析的抗体组学研究,一方面可建立抗体谱和疾病发病机制的关系,发现用于诊断的自身抗体标志物;另一方面通过关联分析基因和抗体谱的特征,以揭示遗传、免疫和表型之间的复杂关系。上海抗码芯瑞生物科技有限公司由上海交通大学知名教授领衔创立,基于创新型的生物技术面向生命科学研究提供技术服务。抗码芯瑞具有领先的噬菌体免疫沉淀测序(PhIP-seq)技术平台,并制备了3种ready-to-use的噬菌体展示肽库,包括12-aa随机肽库、HumanScan全覆盖人蛋白质组库和TCGA肿瘤突变库,并且可提供定制化的建库服务。抗码芯瑞可提供基于这些肽库的高通量筛选、数据分析和后续验证等技术服务,为抗体组学研究提供整体解决方案。
抗码芯瑞联系方式:
电话:021-62202128, 400-8868-750
邮箱:info@abcodebio.com
1、 Sergio Andreu-Sánche et al. Phage display sequencing reveals that genetic, environmental,and intrinsic factors influence variation of human antibody epitope repertoire. Immunity,2023 Apr 25:S1074-7613(23)00171-1.
2、 Arno R. Bourgonje et al. Phage-display immunoprecipitation sequencing of the antibody epitope repertoire in inflammatory bowel disease reveals distinct antibody signatures. Immunity,2023 May 4:S1074-7613(23)00185-1.
3、 Qi H et al. Antibody Binding Epitope Mapping (AbMap) of Hundred Antibodies in a Single Run. Mol Cell Proteomics. 2021;20:100059.






